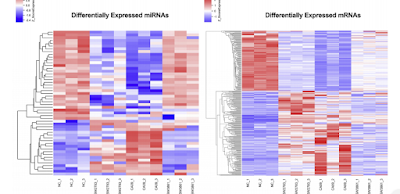
Secuenciación de microarrays
de miARN: Están expresados exclusivamente en los virus con
potencial de capacidad infecciosa humana, determinando la expresión de 4 miRNAs (hsa-miR-96-5p,
hsa-miR-140-5p, hsa-miR-30a-3p y hsa-miR-582-5p) y 5 mRNA relevantes (RCC1,
ERVFRD-1, RANBP1, SCARB2 y RPS29) .
El
microarray de miARN y el análisis cuantitativo por PCR mostraron
que varios miARN, encontró que miR-21-3p que promueve la
virulencia.
BIBLIOGRAFÍA:
- 1. Xia B , Lu J , Wang R , Yang Z , Zhou X , Huang P . Integration analysis of a miRNA-mRNA expression in A549 cells infected with a novel H3N2 swine influenza virus and the 2009 H1N1 pandemic influenza... - PubMed - NCBI [Internet]. Ncbi.nlm.nih.gov. 2019 [cited 31 December 2019]. Available from: https://www.ncbi.nlm.nih.gov/pubmed/31207403
- 2. Xia B , Lu J , Wang
R , Yang Z , Zhou X , Huang
P. miR-21-3p
Regulates Influenza A Virus Replication by Targeting
Histone Deacetylase-8. PubMed - NCBI [Internet].
Ncbi.nlm.nih.gov. 2019 [cited1 1 December 2019]. Available from: https://www.ncbi.nlm.nih.gov/pubmed/29888214